Les tests ADN sur miels, entre promesses et réalités
En apiculture, les tests ADN, en particulier ceux basés sur la métagénomique globale, pourraient aboutir à un véritable « passeport biométrique » des miels, apportant des informations précises, fiables et quasi infalsifiables.
Cet article présente une synthèse des principales méthodes disponibles, en examinant leur précision, leur niveau de développement et leurs limites actuelles.
Quelles technologies derrière les tests ADN sur les miels ?
L’ADN est une molécule remarquablement stable dans le temps, ce qui permet de le retrouver dans des échantillons, même longtemps après la mort des individus ou des cellules qui le composent. Les scientifiques exploitent cette stabilité moléculaire pour analyser les traces laissées par divers organismes dans leur environnement.
Quelle implication cela a-t-il pour l’apiculture ? Au sein d’échantillons complexes comme le miel, on peut s’attendre à détecter l’ADN de multiples organismes : les micro-organismes vivants dans le miel, les abeilles ayant collecté le nectar, les plantes butinées et même, potentiellement, des pathogènes des abeilles ou des végétaux. Ainsi, une simple extraction d’ADN à partir du miel permettrait de révéler un écosystème bien plus vaste que le produit fini ne le laisse supposer.
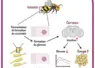
Les extractions d’ADN à partir de miel sont une technique désormais bien maîtrisée dans de nombreux laboratoires, dont l’Inrae à Toulouse. Une fois l’ADN extrait, deux types d’analyses fondamentalement différentes peuvent être réalisés : les approches ciblées « métabarcoding » et les approches globales « métagénomiques ».
Les analyses ciblées (métabarcoding, figure 1) reposent sur l’amplification de courtes régions spécifiques de l’ADN, permettant d’attester de l’identité des espèces présentes. Ces « codes-barres moléculaires » fonctionnent à l’image des codes-barres classiques utilisés en supermarché : un moyen rapide et efficace de scanner un échantillon pour l’identifier.
Concrètement, il s’agit d’amplifier une région précise de l’ADN par la méthode PCR, puis de séquencer le fragment obtenu afin de le comparer à des bases de données de références connues pour ce même code-barres. Quels marqueurs dès lors choisir ? L’ensemble de la communauté scientifique utilise quelques marqueurs génétiques universels, parmi lesquels le 16S pour les bactéries et archées, le COI pour les animaux, l’ITS pour les champignons, et le rbcL, matK ou ITS pour les plantes.
Pour réaliser cette amplification, il est essentiel d’utiliser des amorces universelles, qui sont de petites séquences capables de se fixer sur toutes les molécules d’ADN portant le code-barres ciblé. Ainsi, pour des échantillons complexes comme le miel, l’objectif est d’amplifier l’ensemble des fragments d’ADN correspondant au marqueur recherché : par exemple, le 16S permettra d’identifier toutes les bactéries présentes dans l’échantillon.
Cette approche basée sur les codes-barres moléculaires est notamment celle développée sur les miels par le laboratoire autrichien Sinsoma. Les analyses globales (métagénomique, figure 1) ont pour objectif de séquencer et d’analyser l’ensemble des génomes, et non simplement des petites régions ciblées. Le principe consiste à séquencer l’ensemble des ADN présents dans un échantillon, sans recourir à une amplification PCR.
Prenons l’exemple d’Apis mellifera pour mieux comprendre cette différence. La région du COI, ciblée par l’amplification, mesure environ 400 bases d’ADN, soit moins de 0,0002 % du génome de l’abeille ! Sur des échantillons complexes, l’ensemble des ADN présents, correspondant à tous les organismes, est séquencé simultanément et comparé à des bases de données beaucoup plus vastes, idéalement composées de génomes complets.
Cette approche est appelée métagénomique globale, car elle permet d’analyser simultanément des données génomiques à grande échelle provenant de différentes espèces ; les analyses deviennent beaucoup plus fines et précises. Cette stratégie est notamment développée par l’entreprise estonienne Celvia, ainsi qu’au sein du laboratoire de l’Inrae de Toulouse.
Problématiques apicoles expliquant l’engouement actuel pour les tests ADN
La traçabilité et la lutte contre la fraude alimentaire reposent aujourd’hui sur un large éventail de méthodes. Les standards actuels combinent l’analyse pollinique (mélissopalynologie), l’évaluation de critères physico-chimiques et des analyses chimiques ciblées, telles que le profilage RMN, l’étude des oligosaccharides ou encore l’analyse des ratios isotopiques.
Toutefois, ces approches ne permettent pas toujours de garantir une traçabilité et une authenticité parfaites. En particulier, les miellées issues de plantes peu pollinifères ou contenant des sucres spécifiques présentent un risque élevé de non-conformité dans ces analyses. Un exemple emblématique est celui du miel de lavande, qui représente 12,8 % de la production nationale en 2024, mais dont la caractérisation reste complexe en raison de ces limitations.
Face à ces défis, les tests ADN appliqués aux miels apparaissent comme une alternative prometteuse, offrant une capacité accrue à discriminer les origines géographiques et à évaluer la qualité des miels avec une précision et une fiabilité renforcées (figure 3).
Concernant l’origine géographique des miels, les tests ADN ouvrent de nouvelles perspectives en permettant d’analyser non seulement l’ADN des pollens, mais aussi celui des plantes nectarifères directement présent dans le nectar. Cette approche pourrait également permettre d’identifier des plantes secondaires aux miellées principales, souvent plus locales et donc particulièrement discriminantes pour tracer l’origine géographique des miels.
En ce qui concerne la qualité des miels, les tests ADN pourraient aussi jouer un rôle clé dans la détection des adultérations au sirop. Sans entrer dans les détails méthodologiques, l’ajout de sirop peut induire des modifications plus ou moins importantes des ADN présents dans les échantillons.
Des défis majeurs persistent
Contrairement aux méthodes actuelles, les tests ADN permettraient d’analyser simultanément l’origine botanique et géographique, l’authenticité du produit et bien plus. Le laboratoire à l’Inrae de Toulouse explore activement cette approche, mais ces recherches restent encore expérimentales (figure 2). Derrière les promesses, des défis majeurs persistent : bases de données incomplètes, manque d’échantillons référentiels, validation scientifique insuffisante. Ainsi, bien que prometteuses, ces méthodes ne doivent pas être considérées comme matures. À l’heure actuelle, elles ne remplacent donc pas les analyses mélissopalinologiques et chimiques, qui restent les standards du marché. Il est essentiel que leur développement soit validé par des expertises indépendantes, préservées des influences économiques de certains acteurs, afin d’en garantir la fiabilité et leur pertinence pour la filière apicole.